单细胞基因组测序是如何实现的:从实验到分析,步步解析
随着生命科学研究的微观化,基于细胞群体的研究方法已不适用于某些研究领域(如肿瘤异质性、早期胚胎发育等)。单细胞基因组测序通过在单个细胞水平上进行 测序,解决了用组织样本无法获得不同细胞间的异质性信息或样本量太少无法进行常规测序的难题,为科学家研究单个细胞的行为、机制等提供了新的方向。
【应用概览】
从2011年Navin等人将全基因组扩增技术与高通量测序技术结合起来进行乳腺癌细胞研究开始,单细胞基因组测序技术已逐渐被应用于神经科学、生殖系演 化、器官发生、肿瘤、临床诊断、免疫学、微生物学、组织嵌合、胚胎发育以及产前遗传学诊断等研究领域,并被《自然方法》杂志( Nature Methods )列入2013年度值得期待的技术之一。

单细胞测序发展历程及出版物数量和类目[1]
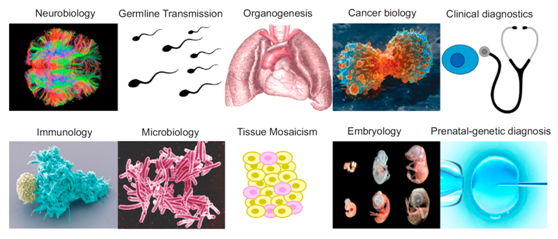
单细胞基因组测序技术被广泛应用于生物科学和生物医学领域[1]
【技术流程】
单细胞基因组测序主要包括四个步骤:单细胞分离→全基因组扩增→高通量测序→数据分析。其中,单细胞分离及全基因组扩增对最终结果的准确性起到了关键作用。

单细胞测序流程示意图[2]
1. 单细胞分离
将单个细胞从组织中分离出来是单细胞基因组测序的第一步。虽然已有较成熟的用于分离在细胞群体中占多数的单细胞的技术,但分离在细胞群体中罕见 (<1%)的单细胞仍是一项艰巨的挑战。目前常用的单细胞分离方法主要有:毛细管吸取、流式细胞分选术、激光捕获显微切割技术(LCM)、微流控技 术等。
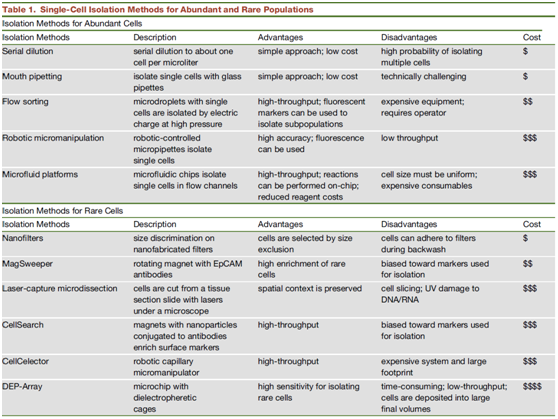
单细胞分离技术[1]
2. 全基因组扩增(WGA)
由于单细胞中DNA的含量非常小(约6pg/细胞),达不到测序仪的检测要求,故在测序前,必须先对单细胞中的DNA进行扩增富集。现有的全基因组扩增方 法按原理可分为三类:基于PCR的WGA方法、多重链置换扩增(MDA)、多次退火环状循环扩增技术(MALBAC)。
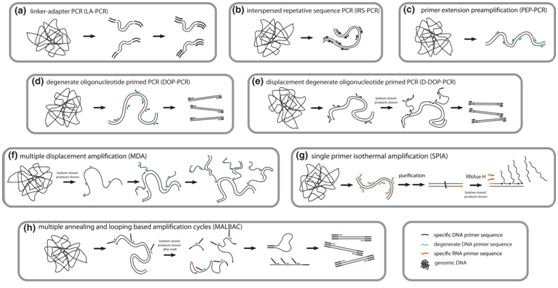
几种主要的全基因组扩增技术原理示意图[3]
1) 基于PCR的WGA方法
基于PCR的WGA方法有很多种,如LA-PCR、PEP-PCR、DOP-PCR等。目前常用的是DOP-PCR和D-DOP-PCR,两者都是通过加 入随机引物与模板结合的方式来达到扩增整个基因组的目的,不同之处主要在于后者所用的DNA聚合酶具有链置换功能。此类方法常见的商业试剂盒 为:GenomePlex Single Cell Whole Genome Amplification Kit (Sigma-Aldrich, USA)和PicoPLEX WGA Kit (Rubicon Genomics, USA)。
2) MDA
MDA是一种滚环扩增技术,其原理主要是先让DNA变性成单链,加入随机引物与模板退火结合,再通过φ29 DNA聚合酶或Bst DNA聚合酶进行链置换合成反应使DNA扩增。标准的MDA实验方案中采用的是φ29 DNA聚合酶,因为它相对Bst DNA聚合酶有更高的产量、保真性以及更低的扩增偏好性。其常见试剂盒为:REPLI-g Single Cell Kit (Qiagen, Germany)。
3) MALBAC
MALBAC通过特殊的随机引物,使扩增产物首尾互补成环,进行近乎线性的全基因组预扩增,再通过PCR技术进行指数式扩增,从而降低扩增过程中错误累 积。其常见试剂盒为:MALBAC Single Cell WGA Kit (Yikon Genomics, China)。
3. 高通量测序
自2005年454 Life Sciences公司(2007年被Roche正式收购)推出454 FLX焦磷酸测序平台以来,高通量测序平台不断更新换代,但目前获得国家食品药品监督管理总局(CFDA)批准可用于临床应用的二代测序仪只有国内少数几 家测序服务公司的Ion Proton(Life Technologies)、NextSeq 500(Illumina)等测序平台。
Ion Proton和NextSeq 500都是采用边合成边测序(SBS)的测序原理,不同之处在于Ion Proton是以dNTP水解时释放H+所导致的局部酸碱度变化为检测信号,而NextSeq 500是以dNTP携带的荧光信号为检测信号。
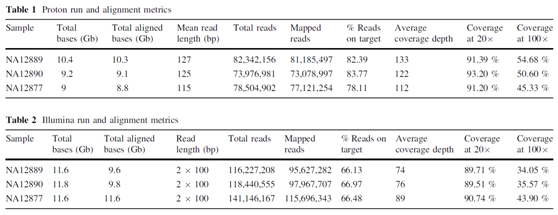
Ion Proton和Hiseq平台测序结果比较[4]
4. 数据分析
数据分析基本流程

目前常见的变异类型为单核苷酸多态性(single nucleotide polymorphism, SNP)、插入缺失标记(insertion-deletion, InDel)和拷贝数变异(copy number variation, CNV)。
1) SNP、InDel
SNP和InDel是基因组上单个点或若干个(通常不大于30个)点上碱基的改变,在肿瘤、胚胎植入前遗传学诊断(PGD)等领域较常见。目前大多数检测 效果较好的分析软件都是先利用Genome Analysis Toolkit(GATK)初步检测SNP或INDEL,再根据不同项目需求对检测到的SNP和INDEL进行筛选,最后通过公共数据库或本地数据库对 SNP和INDEL进行注释。
2) CNV
CNV是一种基因组结构变异(SV),是由基因组发生重排而导致的基因组大片段(一般长度在1 kb 以上)拷贝数增加或者减少。CNV检测目前在肿瘤发生及演化、神经元异质性研究、胚胎植入前筛查(PGS)等领域应用广泛,但其检测方法有很多,原理和流 程也不尽相同。以目前热门的PGS产品为例,CNV的检测流程主要包括:将每条染色体分割为20kb连续的窗口→统计待测样本序列中比对到各窗口中的唯一 比对片段数→数据标准化→计算检测指标→寻找断点→筛选拷贝数变异。
参考文献
1. Wang Y, Navin Nicholas E. Advances and Applications of Single-Cell Sequencing Technologies. Molecular Cell.58(4):598-609.
2. Liang J, Cai W, Sun Z. Single-Cell Sequencing Technologies: Current and Future. Journal of Genetics and Genomics. 2014;41(10):513-28.
3. Blainey PC. The future is now: single-cell genomics of bacteria and archaea. FEMS Microbiology Reviews. 2013;37(3):407-27.
4. Boland JF, Chung CC, Roberson D, Mitchell J, Zhang X, Im KM, et al. The new sequencer on the block: comparison of Life Technology’s Proton sequencer to an Illumina HiSeq for whole-exome sequencing. Human Genetics. 2013;132(10):1153-63.

 推荐书籍
推荐书籍





